Proteine sind in allen lebenden Zellen enthalten und für die Aufrechterhaltung der Körperfunktionen unerlässlich. Sie bestehen hauptsächlich aus Aminosäuren und ermöglichen als Katalysatoren (Enzyme) biochemische Reaktionen, die ansonsten nicht ablaufen würden. Enzyme steuern zum Beispiel die Verdauung und das Immunsystem. „Welche biochemischen Reaktionen ablaufen und wie, hängt von der Struktur der Proteine ab“, sagt Prof. Dr. Olav Schiemann vom Institut für Physikalische und Theoretische Chemie der Universität Bonn. Proteine sind nicht starr, sondern können über bewegliche „Scharniere“ ihre Form verändern. Dieses Wechselspiel zwischen Struktur und Dynamik entscheidet darüber, was passiert. Enzym und umzusetzende Substanz müssen wie Schlüssel und Schloss zusammenpassen, damit ein bestimmter Vorgang katalysiert wird.
YopO ist in der Membran verankert und dadurch besonders stabil
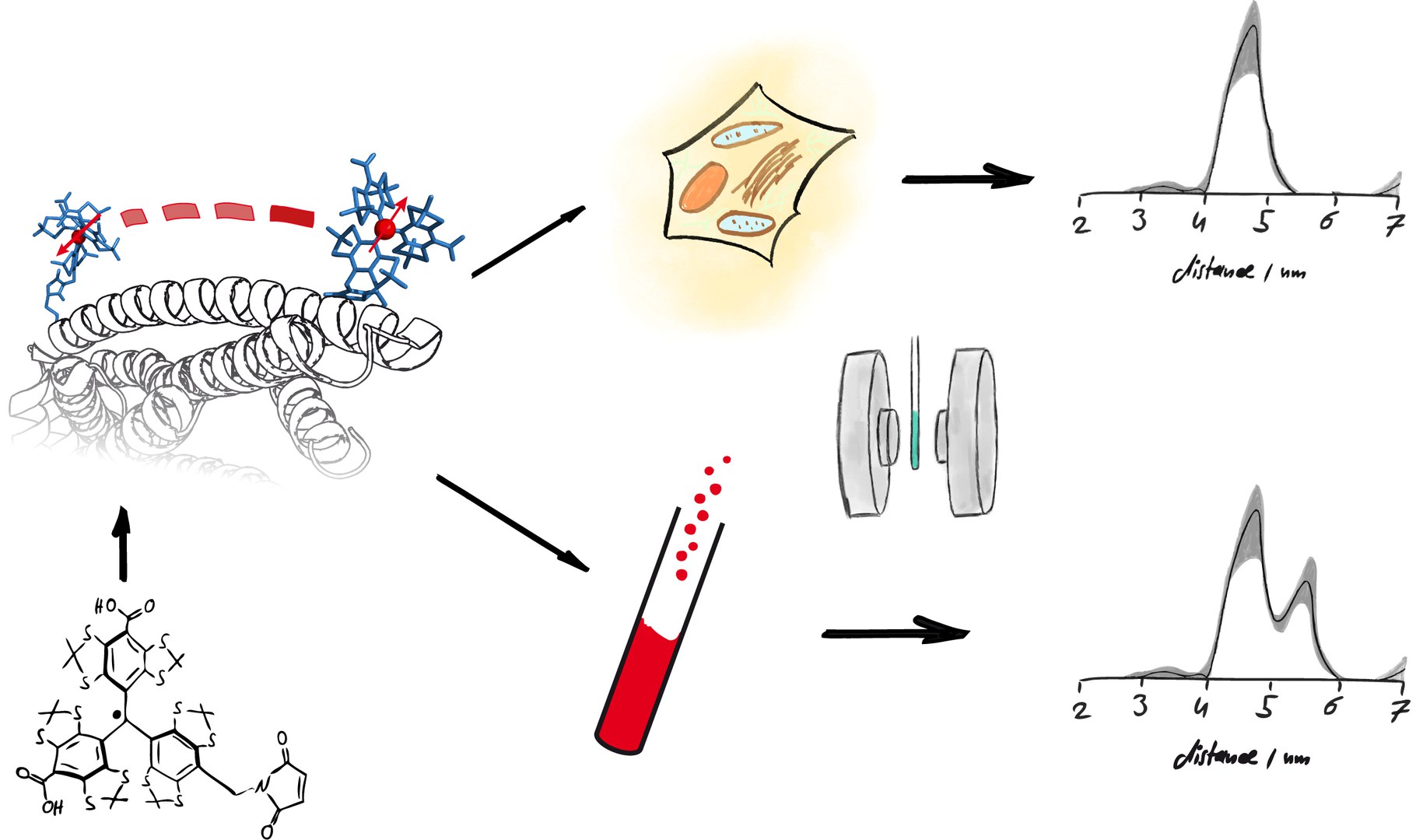
Die Wissenschaftler verwendeten für ihre Untersuchung ein Protein von Pesterregern (Yersinien). Diese tricksen das Immunsystem aus, indem sie Proteine wie zum Beispiel YopO (Yersinia outer protein O) in die angreifenden Fresszellen spritzen. YopO bindet an das Aktin der Abwehrzellen, worauf die Immunzellen die Erreger nicht mehr umschließen und verdauen können. „Wir nutzten YopO, weil dieses Enzym medizinisch interessant ist und in einer Membran verankert beziehungsweise immobilisiert werden kann“, erläutert Schiemann. „Letzteres ist eine wichtige Voraussetzung für unsere Messungen bei Raumtemperatur.“
Nico Fleck aus Schiemanns Forschungsgruppe entwickelte hierfür speziell auf Untersuchungen in der Zelle abgestimmte Spinlabel. Dabei handelt es sich um winzige „Fähnchen“, die Teammitglied Caspar A. Heubach an unterschiedliche Positionen des Proteins anbrachte. Mit der DQC (Double Quantum Coherence)-Methode, die wie ein Lineal auf molekularer Ebene funktioniert, vermaß Tobias Hett aus der Gruppe anschließend die Abstände zwischen den Fähnchen. „Wenn wir die Abstände zwischen den Spinlabeln kennen, können wir darauf schließen, welche Strukturen ein bestimmtes Enzym einnehmen kann“, sagt Hett. Das funktioniert ähnlich einem „Navi“ für Moleküle, weil auch das Orientierungssystem für Fahrzeuge auf Abstandsmessungen beruht.
Die Forscher führten die DQC-Methode an YopO im Reagenzglas und zum Vergleich in Eizellen des Afrikanischen Krallenfrosches durch, die als Modellorganismen in der Wissenschaft häufig verwendet werden. Für die Messungen in der Zelle wurde das mit den Fähnchen markierte YopO in die Eizellen mit einer Spritze injiziert, „sehr ähnlich wie die Pesterreger es auf molekularer Ebene auch tun“, erklärt Nico Fleck. Dabei zeigte sich, dass YopO in wässriger Lösung im Reagenzglas eine größere Zahl verschiedener Strukturen einnehmen konnte als in den Eizellen. „Im Reagenzglas ist YopO strukturell beweglicher als in lebenden Zellen“, sagt Schiemann. „In den Zellen engen Strukturen wie zum Beispiel Membranen und Interaktionen mit anderen Proteinen die Strukturvielfalt von YopO ein.“
Grundlegendes Prinzip
Das ist ein Befund nicht nur für YopO, sondern er gilt als grundlegendes Prinzip: Im Reagenzglas gibt es kein „Korsett“ durch andere Zellstrukturen, die Entfaltungsmöglichkeiten für Enzyme sind größer. Das hat nach den Erkenntnissen der Wissenschaftler Konsequenzen für sämtliche Untersuchungen mit Biomolekülen. „Untersuchungen an den isolierten Biomolekülen sind sicherlich essentiell. Für ein vollständiges Bild sollte man solche Strukturen und Dynamiken aber unter so natürlichen Bedingungen wie möglich untersuchen“, sagt Schiemann. Caspar Heubach ergänzt: „Wenn sich die Aussagen einer Studie auf biomolekulare Vorgänge in Zellen beziehen, sollte man, wie hier, die Struktur und Dynamik der Proteine auch in lebenden Zellen untersuchen.“
Ergebnisse sind für die Pharmaforschung interessant
Da Proteine verschiedene Zellvorgänge steuern, stehen sie auch bei der Suche nach neuen Therapien im Fokus. Die Ergebnisse des Forschungsteams der Universität Bonn sind deshalb auch potenziell interessant für die Pharmaforschung, sind die Wissenschaftler überzeugt. „Die Wechselwirkungen in der Zelle sind wichtig für die Struktur und Dynamik der Proteine“, sagt Schiemann. „Es macht deshalb einen Unterschied, wie die Struktur von Enzymen für die Wirkstoffsuche bestimmt wird.“
Publikation: Nico Fleck, Caspar A. Heubach, Tobias Hett, Florian R. Haege, Pawel P. Bawol, Helmut Baltruschat, Olav Schiemann: SLIM: A short-linked, highly redox-stable trityl label for high sensitivity in cell EPR distance measurements, Angewandte Chemie, DOI: 10.1002/anie.202004452
Kontakt für die Medien:
Prof. Dr. Olav Schiemann
Institut für Physikalische und Theoretische Chemie
Universität Bonn
Tel. 0228/732989
E-Mail: schiemann@pc.uni-bonn.de