Ustilago maydis befällt die oberirdischen Teile der Maispflanze und vermehrt sich in ihnen. Am Ort der Infektion bilden sich oft gewaltige tumorähnliche Gewebewucherungen. Diese sogenannten Gallen können die Größe eines Kinderkopfes erreichen. Auslöser der Wucherungen sind vom Pilz ausgeschüttete Moleküle, Effektoren genannt. Sie manipulieren den Stoffwechsel der Pflanze und unterdrücken ihr Immunsystem. Zudem fördern sie im Mais Zellwachstum und -teilung. Sie greifen dazu in einen Signalweg der Pflanze ein, der durch das pflanzliche Hormon Auxin reguliert wird.
„Der Pilz nutzt diesen Auxin-Signalweg für seine Zwecke“, erklärt Prof. Dr. Armin Djamei, der am INRES-Institut der Universität Bonn die Abteilung Pflanzenpathologie leitet. „Denn das massive Wachstum des Gewebes verschlingt Energie und Ressourcen, die dann für die Verteidigung gegen Ustilago maydis fehlen. Zudem findet der Pilz in den Wucherungen ein ideales Nährstoffangebot vor und kann sich dort gut vermehren.“ Die Bildung der charakteristischen Gallen ist daher durchaus im Interesse des Krankheitserregers.
„Wir wollten deshalb herausfinden, wie er diese Wucherungs-Prozesse fördert“, sagt Djamei. „Wir haben dazu nach Erbanlagen des Pilzes gefahndet, die ihn befähigen, den Auxin-Signalweg seiner Wirtspflanze und damit ihr Zellwachstum zu steuern.“ Begonnen hat die aufwändige Suche vor sieben Jahren am Gregor Mendel Institut in Wien. Später setzte der Nutzpflanzenforscher die Arbeiten am Leibniz-Institut in Gatersleben und zuletzt an der Universität Bonn fort.
Erreger programmiert seinen Wirt um
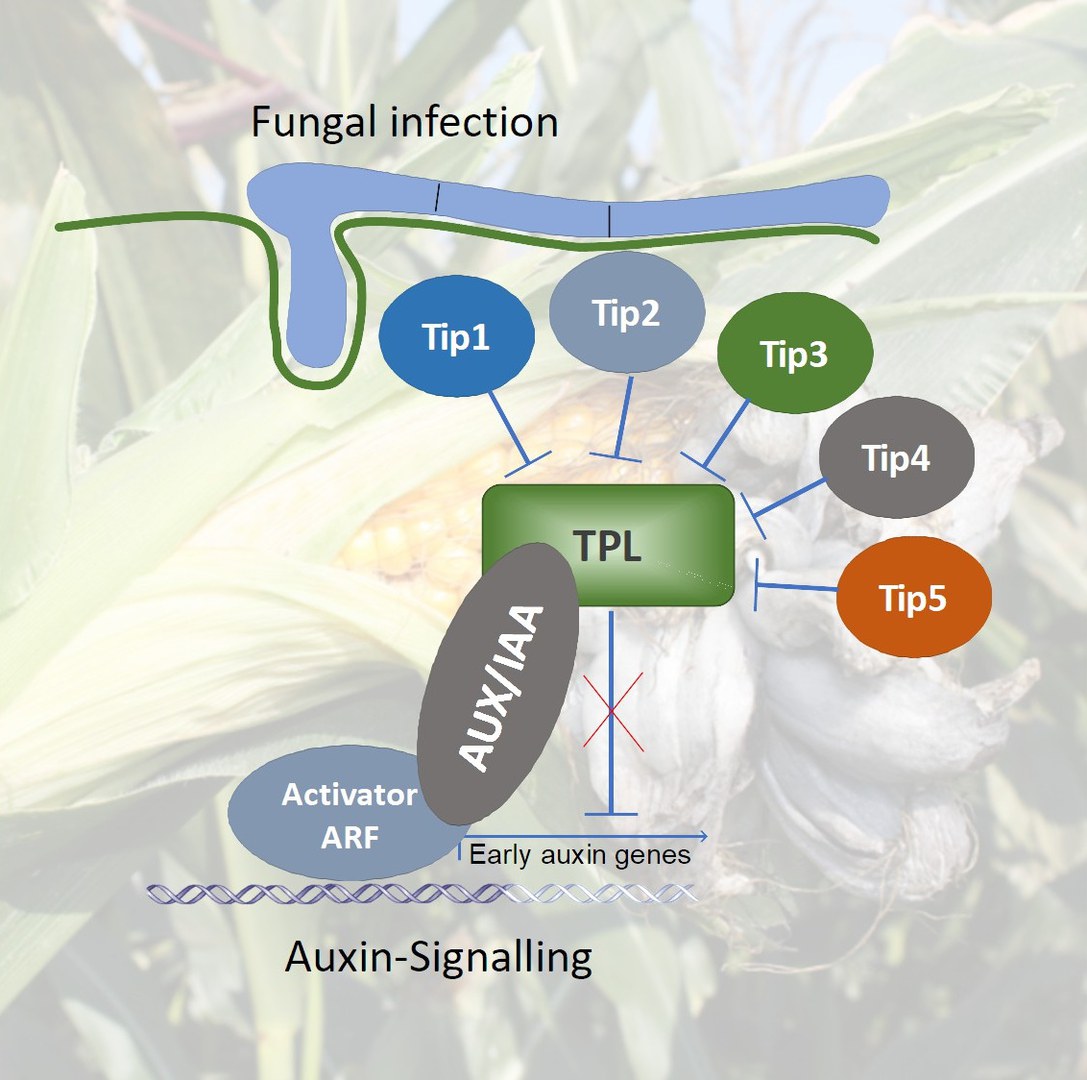
Mit Erfolg: Zusammen mit seinen Mitarbeiterinnen und Mitarbeitern konnte er fünf Gene identifizieren, mit denen der Pilz den Auxin-Signalweg der Wirtspflanze manipuliert. Diese fünf Erbanlagen, Tip1 bis Tip5 genannt, bilden ein sogenanntes Cluster: Wenn man sich das gesamte Erbgut von Ustilago maydis als ein dickes Lexikon vorstellt, liegen die fünf darin gewissermaßen auf aufeinanderfolgenden Seiten.
Gene sind Bauanleitungen – der Pilz benötigt sie, um bestimmte Moleküle herzustellen. „Die von den fünf Tip-Erbanlagen produzierten Moleküle können an ein Protein der Maispflanze binden, das in der Fachwelt unter dem Namen Topless bekannt ist“, erklärt Dr. Janos Bindics. Der ehemalige Mitarbeiter des Gregor Mendel Instituts hat zusammen mit seiner Kollegin Dr. Mamoona Khan viele der zentralen Experimente der Studie durchgeführt.
Topless ist eine zentrale Schaltstelle, die sehr verschiedene Signalwege in der Pflanze unterdrückt. Die Pilzeffektoren, die von den fünf Tip-Genen produziert werden, heben diese Unterdrückung auf – und zwar ganz spezifisch für Signalwege, die dem Pilz nützen, beispielsweise den Auxin-gesteuerten Wachstumssignalweg. Andere von Topless kontrollierte Signalwege werden dagegen nicht beeinflusst. „Der Pilz agiert bildlich gesprochen mit chirurgischer Präzision“, betont Djamei. „Er erreicht exakt, was er erreichen muss, um die Maispflanze bestmöglich infizieren zu können.“
Erkenntnisse für die Grundlagenforschung
Es gibt eine ganze Reihe von Krankheitserregern, die in den Auxin-Signalweg der von ihnen befallenen Wirte eingreifen. Wie genau, ist oft nicht vollständig verstanden. Möglicherweise spielt Topless dabei aber auch in anderen Nutzpflanzen eine wichtige Rolle. Denn das Protein ist schon vor vielen hundert Millionen Jahren entstanden und hat sich seitdem in seiner zentralen Rolle kaum verändert. Es existiert daher nicht nur in Mais, sondern in ähnlicher Form auch in allen anderen Landpflanzen. Die Forschenden konnten zum Beispiel zeigen, dass die Tip-Effektoren von Ustilago maydis auch in den Auxin-Signalweg anderer Pflanzenarten eingreifen.
Die Erkenntnisse könnten daher dazu beitragen, die Infektions-Prozesse bei wichtigen Pflanzenkrankheiten besser zu verstehen. Insbesondere sind die Ergebnisse für die Grundlagenforschung interessant: „Durch sie wird es erstmals möglich, ganz gezielt bestimmte Effekte des Auxin-Signalwegs zu beeinflussen und so die Wirkung dieser wichtigen Pflanzenhormone noch genauer aufzuklären“, hofft Armin Djamei, Mitglied im Transdisziplinären Forschungsbereich „Sustainable Futures“ und Exzellenzcluster PhenoRob der Universität Bonn.
Förderung und beteiligte Institutionen:
Die Studie wurde durch den Europäischen Forschungsrat (ERC), den Fonds zur Förderung der wissenschaftlichen Forschung (FWF) Österreichs, die Österreichische Akademie der Wissenschaften und die Deutsche Forschungsgemeinschaft (DFG) gefördert. Neben der Universität Bonn waren an den Arbeiten das Gregor Mendel Institut für Molekulare Pflanzenbiologie (GMI) der Österreichischen Akademie der Wissenschaften, das Leibniz-Instituts für Pflanzengenetik und Kulturpflanzenforschung in Gatersleben sowie die Universität Köln beteiligt.
Publikation: Janos Bindics, Mamoona Khan, Simon Uhse, Benjamin Kogelmann, Laura Baggely, Daniel Reumann, Kishor D. Ingole, Alexandra Stirnberg, Anna Rybecky, Martin Darino, Fernando Navarrete, Gunther Doehlemann & Armin Djamei: Many ways to TOPLESS – manipulation of plant auxin signalling by a cluster of fungal effectors; New Phytologist; https://doi.org/10.1111/nph.18315